Les protéines sont présentes dans toutes les cellules vivantes. Elles font partie des structures biologiques constitutives de la vie, car elles portent les instructions permettant aux cellules de remplir leurs fonctions. L’ensemble des protéines d’un organisme se nomme « protéome ». Là où le génome reste relativement fixe, avec une séquence génétique codant les bases de la vie, le protéome est dynamique, se modifiant en fonction des conditions environnementales internes ou externes aux cellules.
Étudier ce protéome et modéliser les dizaines de milliers de protéines qui constituent un organisme revient à étudier les mécanismes du vivant. Sauf que ces structures sont faites d’acides aminés, qui se combinent entre eux, repliant chaque protéine dans une configuration spécifique. Vous pouvez imaginer les protéines comme des sortes d’origamis, en vous représentant chaque petite face pliée comme étant un acide aminé. Mais à l’échelle du vivant, le nombre de combinaisons possibles est énorme ; l’organisme humain est susceptible de voir apparaître des centaines de milliers de protéines. Prédire les repliements, et donc la structure des protéines, est un travail de titan pour les scientifiques. Quelques pour cent du protéome humain ont été décryptés jusqu’ici, autour de 170 000 protéines.
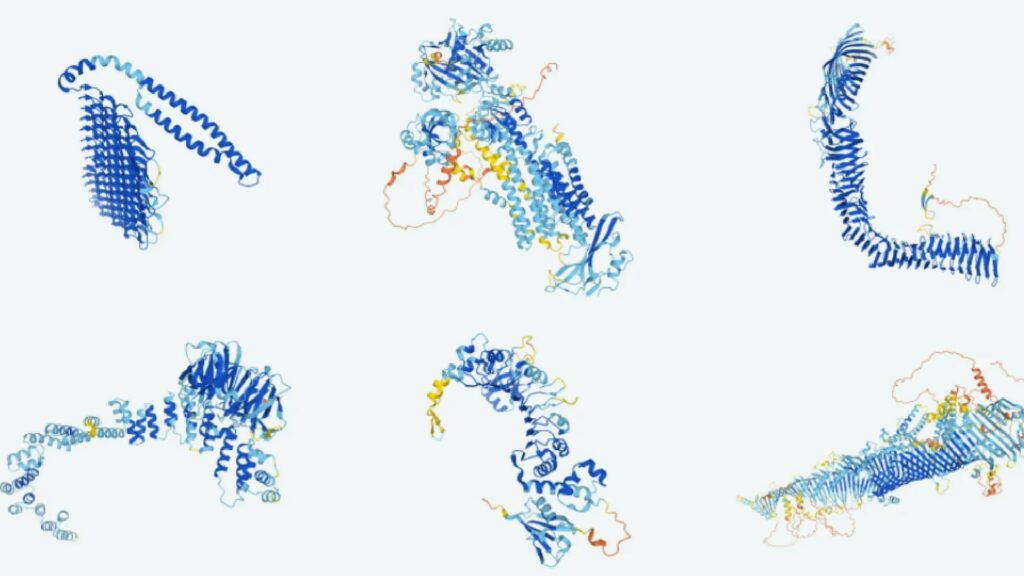
Les protéines peuvent prendre plein de formes différentes en fonction de leurs pliages. Les petites bouclettes colorées sont des acides aminés sur cette image.
Source : DeepMind
Intervient alors l’intelligence artificielle pour prédire la structure complète, en 3D, des protéines. L’entreprise britannique DeepMind développe, depuis 2018, un algorithme de machine learning, AlphaFold, destiné à la prédiction des repliements de protéines. En décembre 2020, AlphaFold atteignait une précision annoncée comme inédite. Le 15 juillet 2021, DeepMind publiait enfin, dans Nature, le code source et le fonctionnement de ce système. Quelques jours plus tard, le 22 juillet, ce sont les résultats obtenus grâce au logiciel lui-même qui ont été publiés, toujours dans Nature.
Cela aboutit à une base de données contenant les structures prédites de 350 000 protéines de l’organisme humain, auxquelles s’ajoutent 20 protéines d’autres organismes (comme la levure ou la mouche du vinaigre). C’est plus du double des protéines dont la structure a déjà été prédite. « Ce qui nous a pris des mois et des années à faire, AlphaFold a pu le faire en un week-end. J’ai le sentiment que nous venons de faire un bond d’au moins un an par rapport à la situation d’hier », a confié le professeur John McGeehan, dans une série de commentaires indépendants dédiés à cette étude.
Un catalogue de 350 000 protéines
Pour parvenir à un résultat aussi étendu, soit 44 % des protéines constituant tout le protéome humain et 60 % des acides aminés codés par notre génome, l’équipe de recherche a entraîné l’algorithme à partir des 170 000 structures protéiques déjà connues. L’algorithme a appris à prédire la forme d’une protéine… à partir de ses acides aminés.
Car, oui, identifier la séquence d’acides aminés d’une protéine est la phase la plus « simple », cela ne suffit pas : il faut modéliser sa structure en 3D. La forme que prend la protéine, c’est-à-dire la façon dont elle se replie comme nos origamis, détermine très largement la fonction qu’elle remplit — un peu à la manière d’une fourchette ou d’un couteau, dont vous pouvez déterminer l’utilité à partir de sa forme.
Ainsi, en couvrant près de 60 % de tous les acides aminés codés dans l’organisme humain, AlphaFold a pu déterminer la structure de 44 % environ de toutes nos protéines.
Ce n’est pas tout. Dans les travaux publiés, les auteurs relèvent qu’AlphaFold a mis en évidence l’existence de nombreuses protéines humaines « désordonnées », expliquant que l’algorithme n’ait pas pu en prédire la structure. Ces protéines n’adoptent pas une forme unique, mais variable. Elles peuvent éventuellement adopter une forme fixe lorsqu’elles se lient à une partenaire.
Comment fonctionne la vie (et autres questions)
Cette base de données d’une ampleur inédite — un catalogue de 350 000 protéines — est disponible en ligne. Tous les scientifiques y ont accès, de quoi donner un petit coup d’accélérateur aux recherches. Edith Heard, directrice de l’European Molecular Biology Laboratory avec lequel a collaboré DeepMind, estime que « cela va transformer la compréhension du fonctionnement de la vie ».
Il s’agit d’abord d’amélioration les connaissances fondamentales sur la biologie humaine, végétale et animale. S’ajoute à cela que les recherches sur les protéines sont très actives dans les domaines médicaux et écologiques — notamment parce que certaines protéines permettent de résister aux maladies, tandis que d’autres peuvent participer à une infection. Par exemple, face à la crise sanitaire, une équipe de DeepMind mobilise l’algorithme AlphaFold pour cerner comment les protéines du coronavirus s’attachent aux cellules humaines.
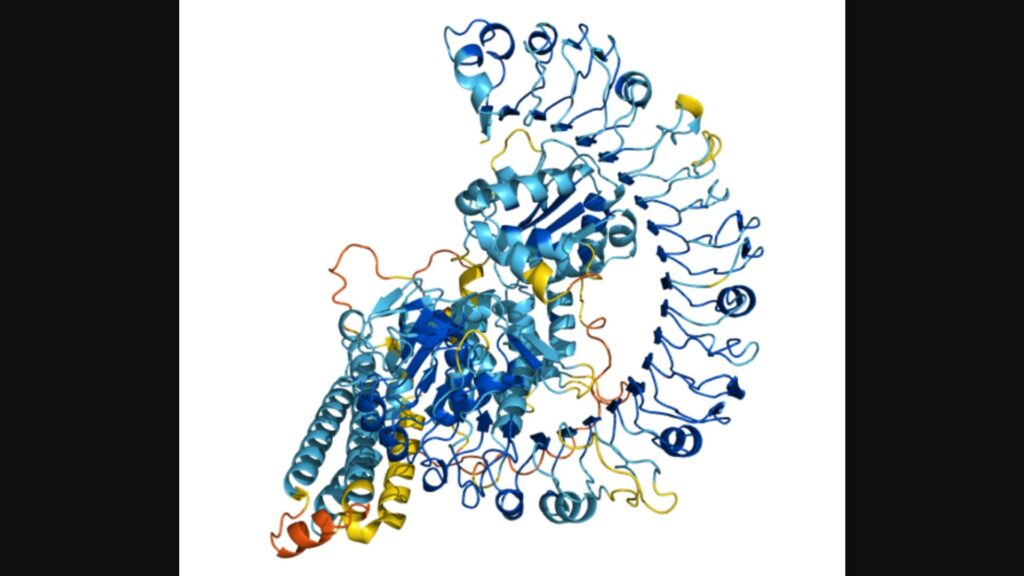
Voici Q8W3K0. Il s'agit d'une protéine qui permet à certaines plantes de résister aux maladies.
Source : DeepMind
Plus largement, les recherches protéomiques (dédiées à l’étude du protéome) visent aussi à créer des médicaments. Parmi les protéines prédites dans le nouveau catalogue développé avec AlphaFold, on trouve les protéines contenues par des organismes tels que le parasite de la malaria ou la bactérie à l’origine de la tuberculose.
Quant à l’avenir des prédictions de protéines réalisées par IA, DeepMind annonce vouloir voir grand. Puisque l’algorithme a appris à déterminer la forme d’une protéine à partir du séquençage de ses acides aminés, les scientifiques veulent rassembler toutes les protéines dont ce séquençage a déjà été réalisé, pour y appliquer AlphaFold. Cela représente 100 millions de structures. Ce champ de recherche fait par ailleurs face à un autre défi, plus complexe encore : quelles protéines interagissent avec quelles protéines, et quelles en sont les conséquences moléculaires.
+ rapide, + pratique, + exclusif
Zéro publicité, fonctions avancées de lecture, articles résumés par l'I.A, contenus exclusifs et plus encore.
Découvrez les nombreux avantages de Numerama+.
Vous avez lu 0 articles sur Numerama ce mois-ci
Tout le monde n'a pas les moyens de payer pour l'information.
C'est pourquoi nous maintenons notre journalisme ouvert à tous.
Mais si vous le pouvez,
voici trois bonnes raisons de soutenir notre travail :
- 1 Numerama+ contribue à offrir une expérience gratuite à tous les lecteurs de Numerama.
- 2 Vous profiterez d'une lecture sans publicité, de nombreuses fonctions avancées de lecture et des contenus exclusifs.
- 3 Aider Numerama dans sa mission : comprendre le présent pour anticiper l'avenir.
Si vous croyez en un web gratuit et à une information de qualité accessible au plus grand nombre, rejoignez Numerama+.
Toute l'actu tech en un clin d'œil
Ajoutez Numerama à votre écran d'accueil et restez connectés au futur !

Marre des réseaux sociaux ? Rejoignez la communauté Numerama sur WhatsApp !

















